内蒙古RNA免疫共沉淀检测RIP-Sequence
RNA结合蛋白免疫沉淀实验(RIP)的注意事项:防止RNA与蛋白非特异性结合:在实验过程中,需要防止RNA与蛋白的非特异性结合,如使用RNA酶抑制剂,避免使用强去污剂等。避免RNA蛋白质结合被破坏:在样品处理和洗涤过程中,避免使用过高或过低的温度和盐浓度,以免破坏RNA与蛋白质的结合。避免外源RNase污染:需要在实验过程中严格避免外源RNase的污染。如使用RNase-free的试剂和耗材,保持实验室的清洁等。抑制内源RNase的活性:在样品处理和存储过程中,需要加入适量的RNase抑制剂,以抑制内源RNase的活性,防止RNA的降解。RIP-qPCR实验是一种用于研究细胞内特定蛋白质与RNA相互作用的技术。内蒙古RNA免疫共沉淀检测RIP-Sequence
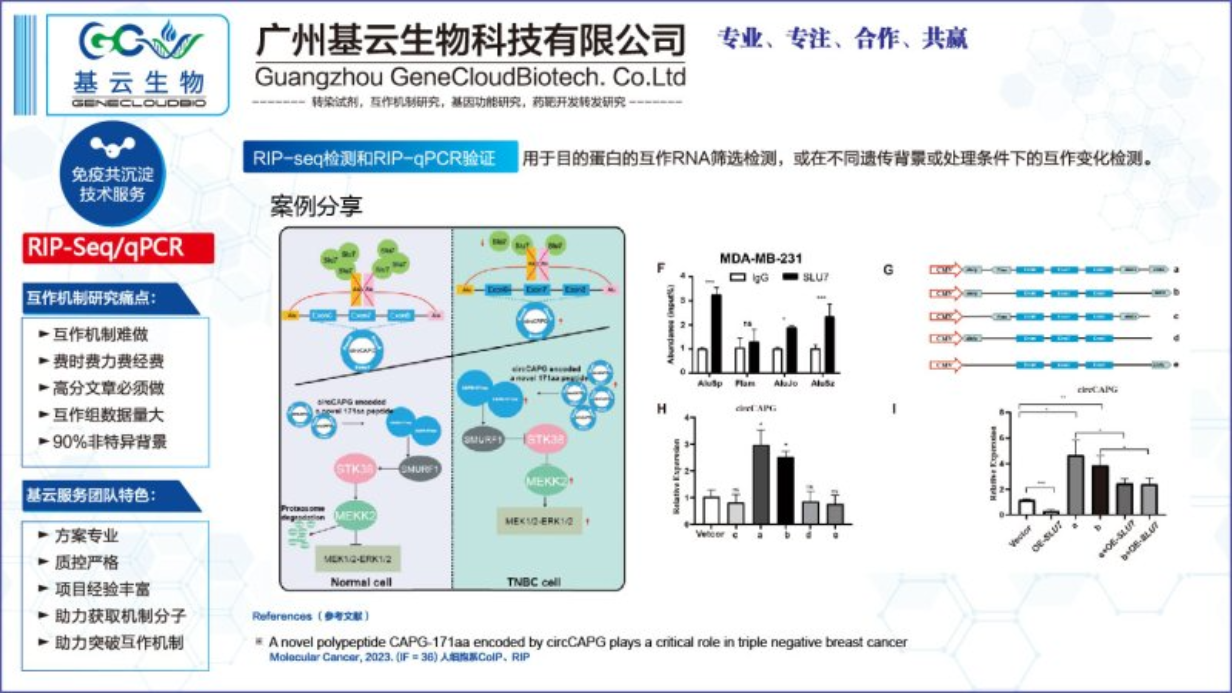
RIP-seq(RNA Immunoprecipitation sequencing)是一种用于研究细胞内RNA与蛋白质结合情况的高通量测序技术。其基本原理是通过目标蛋白的抗体将相应的RNA-蛋白质复合物沉淀下来,然后经过分离纯化,对结合在复合物上的RNA进行高通量测序分析。该技术利用抗体或表位标记物捕获细胞核内或细胞质中的内源性RNA结合蛋白,从而避免非特异性的RNA结合。通过免疫沉淀,RNA结合蛋白及其结合的RNA被一起分离出来。之后,结合的RNA序列通过高通量测序方法进行鉴定和分析。RIP-seq技术结合了免疫沉淀和高通量测序技术,具有高通量、高分辨率和高灵敏度的特点,能够详细、准确地揭示细胞内RNA与蛋白质的相互作用网络。该技术不仅可以用于发现新的RNA结合蛋白和它们的靶标RNA,还可以用于研究RNA在转录后调控、细胞代谢、疾病发生、发展等过程中的功能和机制。总之,RIP-seq技术是一种强大的工具,为研究细胞内RNA与蛋白质的相互作用提供了有力的手段,有助于深入了解细胞内基因表达调控的复杂网络。安徽RIP-Sequencing如果RIP-qPCR实验失败了,该怎么办。
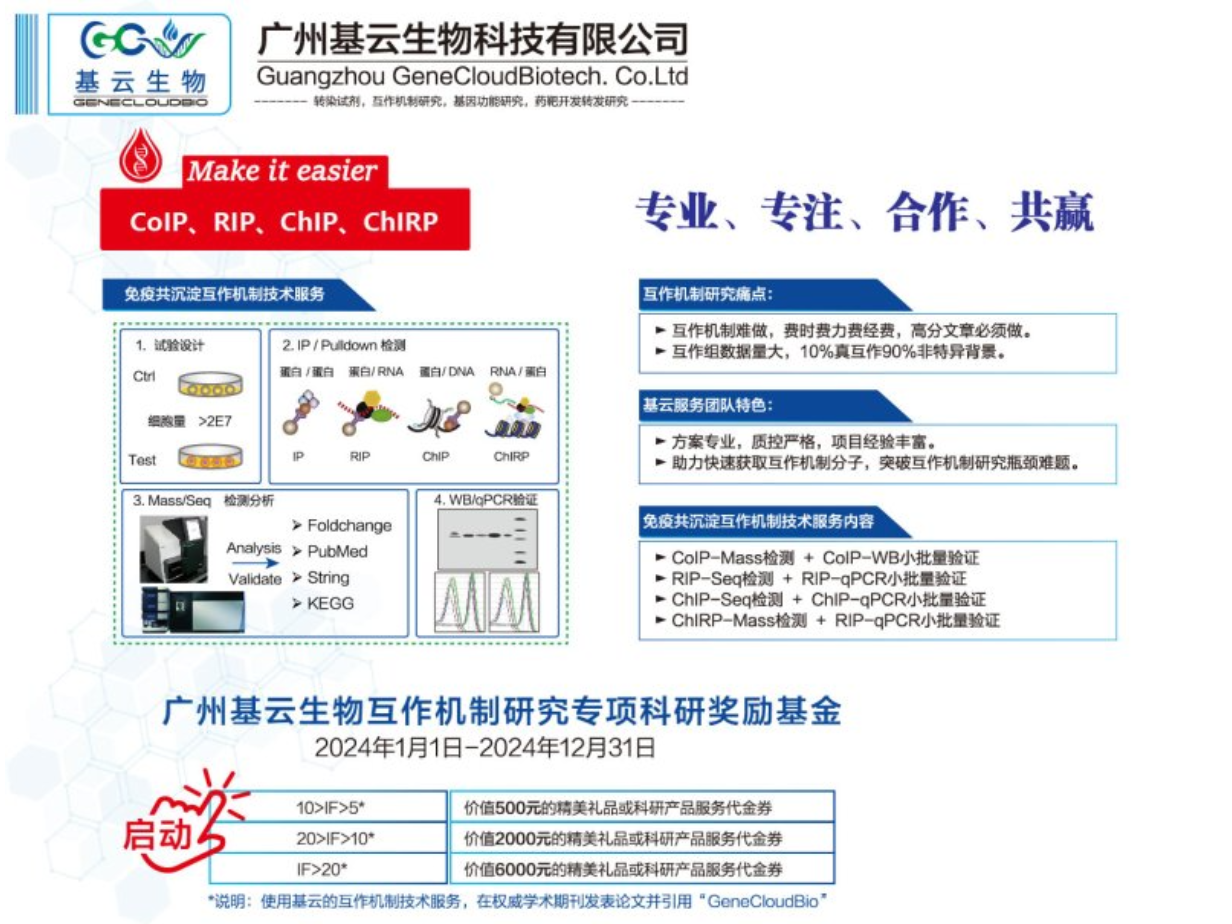
RIP-qPCR实验的基本实验流程如下:细胞裂解:收集目标细胞,使用适当的裂解液进行裂解,释放细胞内的RNA和蛋白质。抗体结合:向细胞裂解液中加入特异性抗体,该抗体能与目标蛋白质结合,形成抗体-蛋白质复合物。免疫共沉淀:加入蛋白A/G磁珠或其他亲和树脂,与抗体-蛋白质复合物结合,然后利用磁力沉淀复合物,去除非特异性结合的蛋白质。RNA提取:从沉淀的复合物中提取RNA,此过程中应使用RNase抑制剂以保护RNA的完整性。逆转录:使用逆转录酶将提取的RNA逆转录为cDNA。qPCR反应:准备qPCR反应液,包括PCR缓冲液、dNTPs、酶、引物和探针等,将cDNA作为模板加入到反应液中,进行qPCR反应。通过反应,可以定量检测与目标蛋白质结合的特定RNA。数据分析:分析qPCR的结果,包括RNA的表达水平和与目标蛋白质的结合强度等。以上流程供参考,实际操作中可能需要根据实验需求进行适当的调整和优化。
RIP-seq实验的基本实验流程如下:准备样本:收集并处理适当的细胞或组织样本,确保样本的质量和数量满足实验需求。免疫沉淀:利用特定蛋白的抗体,通过免疫沉淀技术将RNA-蛋白质复合物从样本中分离出来。这一步骤能够确保只捕获与目标蛋白结合的RNA分子。破碎与文库制备:将捕获的RNA-蛋白质复合物进行破碎处理,释放出RNA分子,并制备成测序文库。这一步骤涉及RNA的纯化和逆转录等过程,以便进行后续的测序分析。高通量测序:利用高通量测序技术对制备好的RNA测序文库进行测序,获得大量的测序数据。这些数据将用于分析RNA与蛋白质的相互作用。数据分析:对测序数据进行生物信息学分析,包括序列比对、峰值调用和注释等步骤,以识别与特定蛋白结合的RNA序列,并揭示它们在细胞内的功能和调控机制。整个实验流程需要严格控制实验条件,确保实验的特异性和准确性。通过RIP-seq实验,可以详细了解RNA与特定蛋白质的相互作用情况,为深入研究基因表达调控和细胞生物学过程提供有力支持。要快速了解RIP实验技术,可以从几个方面入手。

在分子机制研究过程中,RIP-seq(RNA免疫沉淀后测序)实验技术是一种强大的工具,用于详细研究细胞内RNA与蛋白质的相互作用。RIP-seq主要应用于识别和分析与特定RNA结合蛋白(RBP)结合的RNA分子。通过该技术,研究者可以了解RBP在细胞内的靶标RNA,并进一步研究这些RNA在细胞功能、基因表达调控以及疾病发生、发展中的作用。在疾病研究领域,RIP-seq具有广泛的应用。例如,可用于鉴定与疾病相关RBP结合的RNA,从而揭示疾病发生和发展的分子机制。除了疾病研究,RIP-seq还可用于探索细胞内的转录后调控机制。通过分析RBP与RNA的结合模式,可以揭示RNA剪接、修饰、转运和降解等过程中的关键调控因子和机制。此外,RIP-seq还可与其他高通量技术相结合,如转录组测序(RNA-seq)、蛋白质组学等,共同构建细胞内的RNA-蛋白质相互作用网络,为系统生物学研究提供有力支持。总之,RIP-seq实验技术在分子机制研究中具有广泛的应用场景,特别是在疾病相关分子机制、转录后调控机制以及细胞功能研究等方面。随着技术的不断发展,RIP-seq将在分子机制研究领域发挥越来越重要的作用。RIP-seq和RIP-qPCR实验技术有哪些相同点。安徽RNA蛋白互作检测RIP PCR检测
RIP实验通常与哪些实验方法结合使用,以获得更好的研究结果。内蒙古RNA免疫共沉淀检测RIP-Sequence
RIP-qPCR实验的引物设计至关重要,它直接影响到实验的特异性和灵敏度。以下是引物设计的主要要求。特异性:引物应具有高特异性,确保只扩增目标RNA分子,避免非特异性扩增。设计时,应避免与其他基因或RNA存在互补序列。长度与GC含量:引物长度通常在18-25bp之间,GC含量适中(40%-60%),以保证引物的稳定性和退火效率。避免引物二聚体:引物间不应存在互补序列,特别是3’端,以防止引物二聚体的形成。跨内含子设计:对于基因编码区的RNA,引物尽量跨越内含子设计,以避免基因组DNA的污染。3’端修饰避免:引物的3’端不能进行任何修饰,且必须是G或C,因为这两种碱基配对较为稳定,有利于引物的延伸。引物自身互补性:引物自身不应存在互补序列,以避免折叠成发夹结构,影响引物与模板的结合。与模板紧密互补:引物应与模板序列紧密互补,确保PCR的高效扩增。遵循这些要求设计的引物,将大程度提高RIP-qPCR实验的准确性和可靠性。在实验前,还应对设计的引物进行验证,确保其满足实验需求。内蒙古RNA免疫共沉淀检测RIP-Sequence
上一篇: 内蒙古RIP qPCR